シーケンスアセンブリソフトウェア ATGC-MAC は大量のフラグメントの結合処理を高速に行い、
波形の信頼度を考慮してコンセンサス配列の決定をするソフトウェアです。
次世代シーケンサーのデータにも対応しており、ゲノムマッピング、De novo Assembly 等の解析をご利用いただけます。
macOS Monterey 対応
次世代シーケンサー対応機能
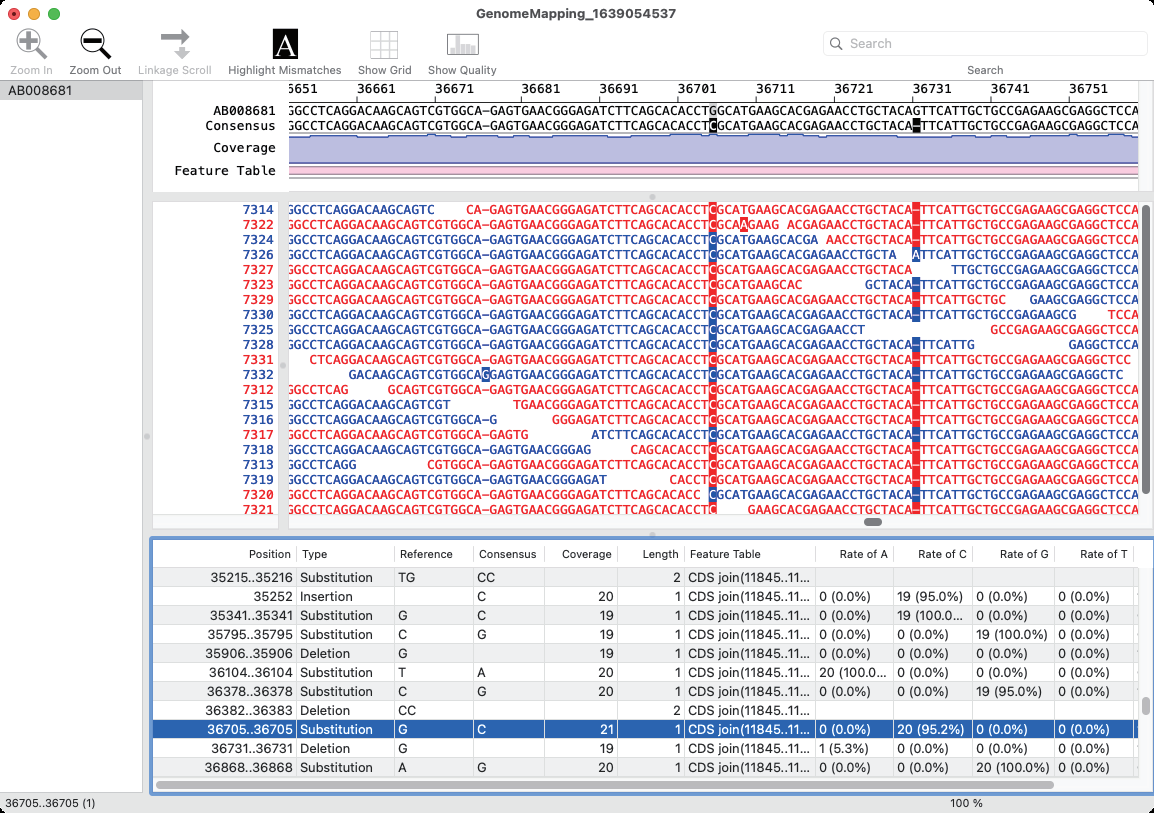
ゲノムマッピング
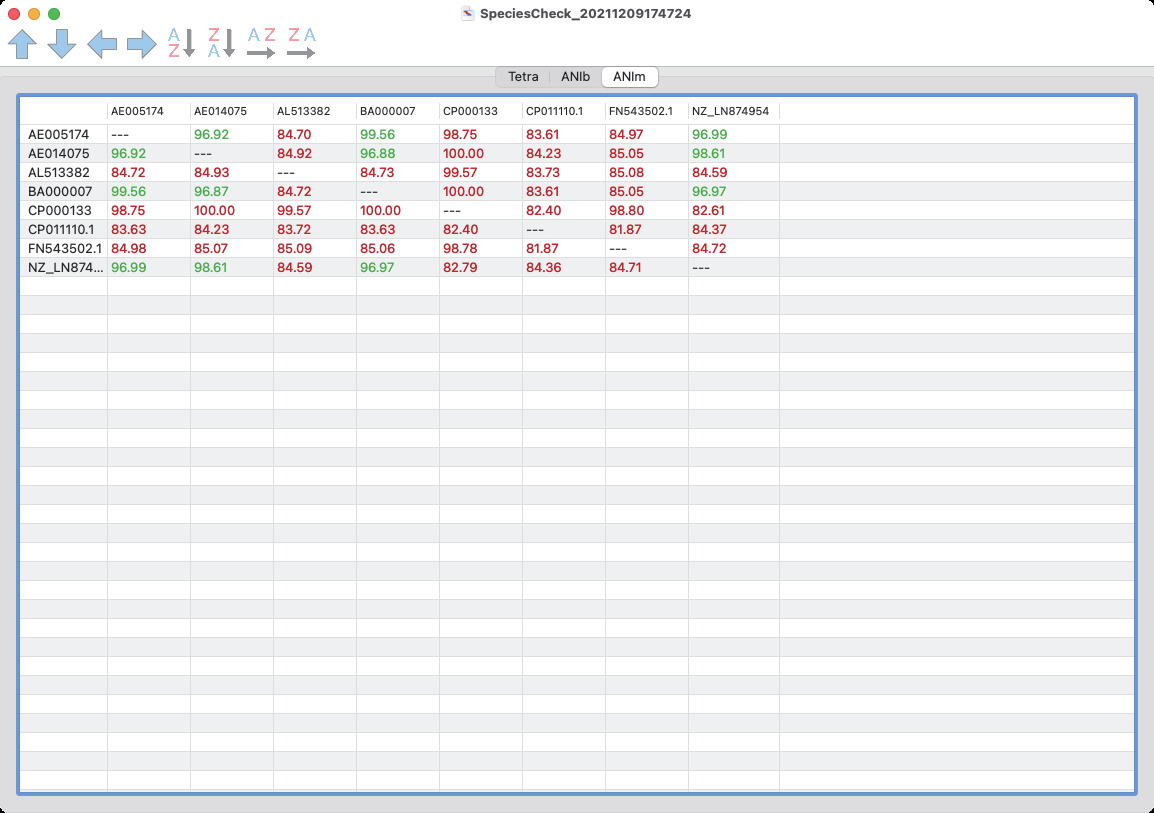
ゲノム種比較
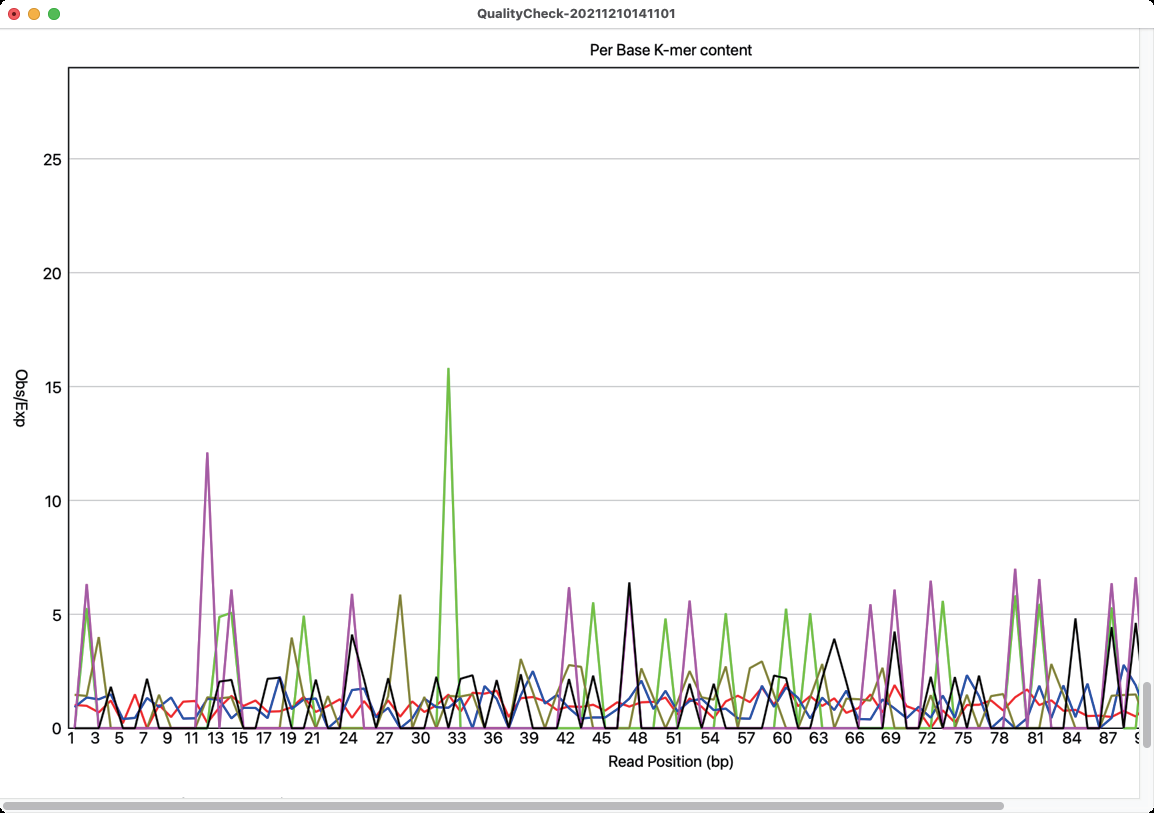
クオリティチェック
高速化 ATGC-MAC Ver.7 と比較して
- ゲノムマッピング (高速化) ● 約10倍 (当社比)
次世代シーケンサーからの断片配列 (FASTA、FASTQ、FNA/QUAL、CSFASTA) を既知のゲノム配列(参照配列)にマッピングします。
- トリミング機能 ●
入力した配列に対してアダプター配列や低クオリティ領域を切り取るトリミング処理を行い新しいファイルに出力します。
- SNP 検出機能 ▲ ●
マッピング結果から SNP の検出を行います。 PCR 重複除去、Realignment およびフィルタ処理を行うことができます。結果は VCF ファイルとして出力することができます。
- ゲノム種比較 (微生物) ▲ ●
Tetra Nucleotide 頻度および ANI (Average Nucleotide Identity) 値を算出して微生物ゲノム間の比較を行います。
- SAM/BAM インポート
マッピング等の解析で一般的に使われる SAM/BAM 形式のファイルを読み込んで、ゲノムマッピングの結果と同時に表示します。
- 変異表
ゲノムマッピングによって得られたコンセンサス配列と参照配列を比較して、その変異を一覧表示します。変異表は印刷や CSV 形式のファイルで出力できます。
- de novo Assembly ●
MIRA ▲ を使用して、次世代シーケンサーから出力された断片配列の de novo Assembly を行います。
- クオリティチェック機能
マッピングやアセンブルなどの解析を行う前にシーケンサーから出力された配列の質 (クオリティスコア、塩基組成、配列長、重複など) を調査します。
- 発現量解析機能 ▲
マッピングの結果を元に発現量解析を行います。
- 配列組成表示機能
任意の範囲を指定してマッピングされた配列の組成を計算して出力します。
- 解析履歴機能
過去に行った解析の結果と条件を表示できます。
シーケンサー出力のアセンブル
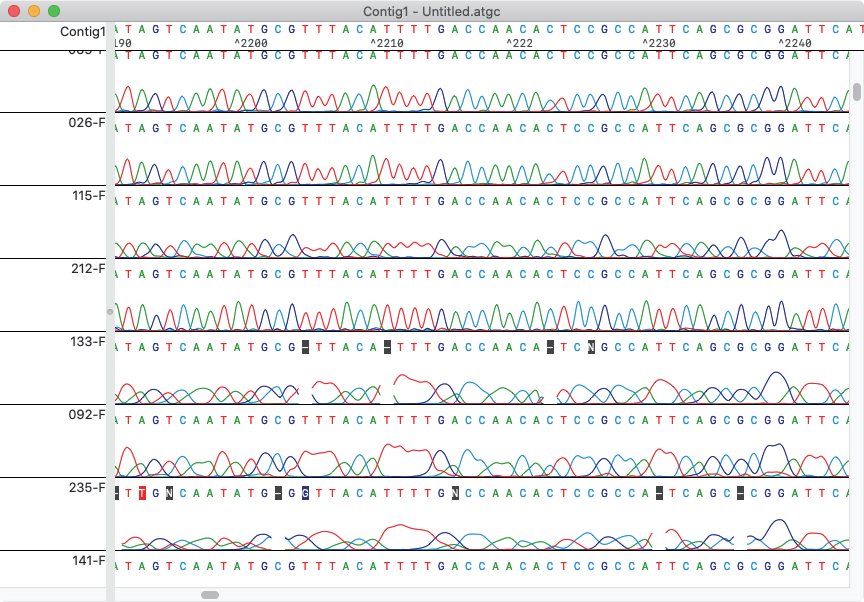
配列・波形同時表示
- 配列・波形同時表示機能 ●
同一画面上に配列ファイルと波形ファイルを表示が可能になりました。
- ベクター配列のトリミング
ベクター配列を検索して、断片配列を読み込む際にその部分を取り除きます。目的の塩基配列のみをアセンブルすることができます。
- アミノ酸翻訳表示
コンセンサス配列からアミノ酸へ翻訳して、コンセンサス配列と同時に表示できます。
- プロジェクトファイル形式の共通化 ●
ATGC で作成したプロジェクトファイルを ATGC-MAC で開けるようになりました。また、その逆も可能です。
 ※Macintosh版の旧バージョンで作成したプロジェクトファイルはMacintosh版のみ対応
※Macintosh版の旧バージョンで作成したプロジェクトファイルはMacintosh版のみ対応
Windows版の旧バージョンで作成したプロジェクトファイルはWindows版のみ対応
動作環境
| 対応機種 | macOS Catalina 以上が動作する機種 |
|---|---|
| 対応 OS | macOS Catalina 以上 |
| メモリ | 4GB 以上 (推奨 8GB 以上) |
| ストレージ容量 | 20GB 以上の空き容量 |
| ハードウェア | 光学ドライブ (インストール時に利用します。) |
| その他 | 本製品のご使用にはインターネットによるライセンス認証が必要 |
●印は Ver.8 で新規追加された機能です。
▲印は別途ダウンロードが必要です。
Copyright © NIHON SERVER CORPORATION All Rights Reserved.








